前言
我认为以节省存储空间为角度出发,Spark作业中的读写压缩文件是必不可少的话题,当然这在MR作业中也有体现和实际解决这种问题,现在我们就要在Spark中解决这种问题。
如果需要安装Lzo可以看我的其他文章
源文件是一份access的原始数据
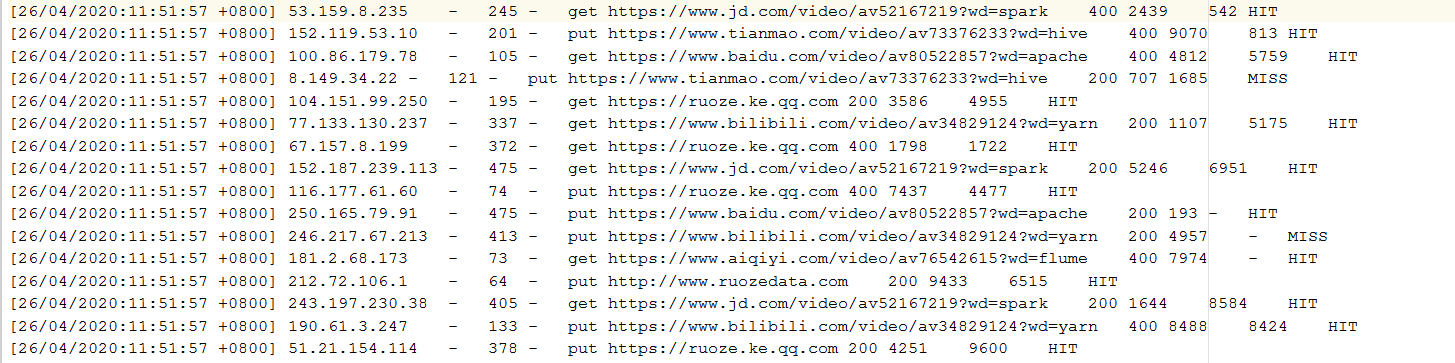
我们在上传到服务器上的时候,使用lzop命令压缩该文件,得到压缩后的文件,上传HDFSF并对该文件创建索引,得到我们代码中需要处理的源文件
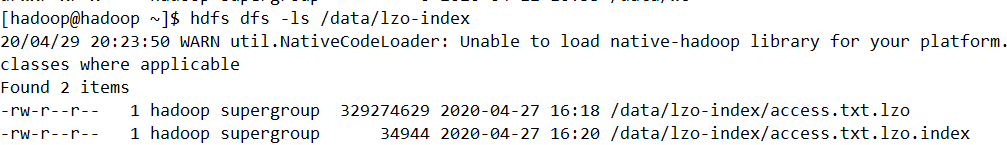
需要注意的是未压缩的文件是1G,压缩后为314M
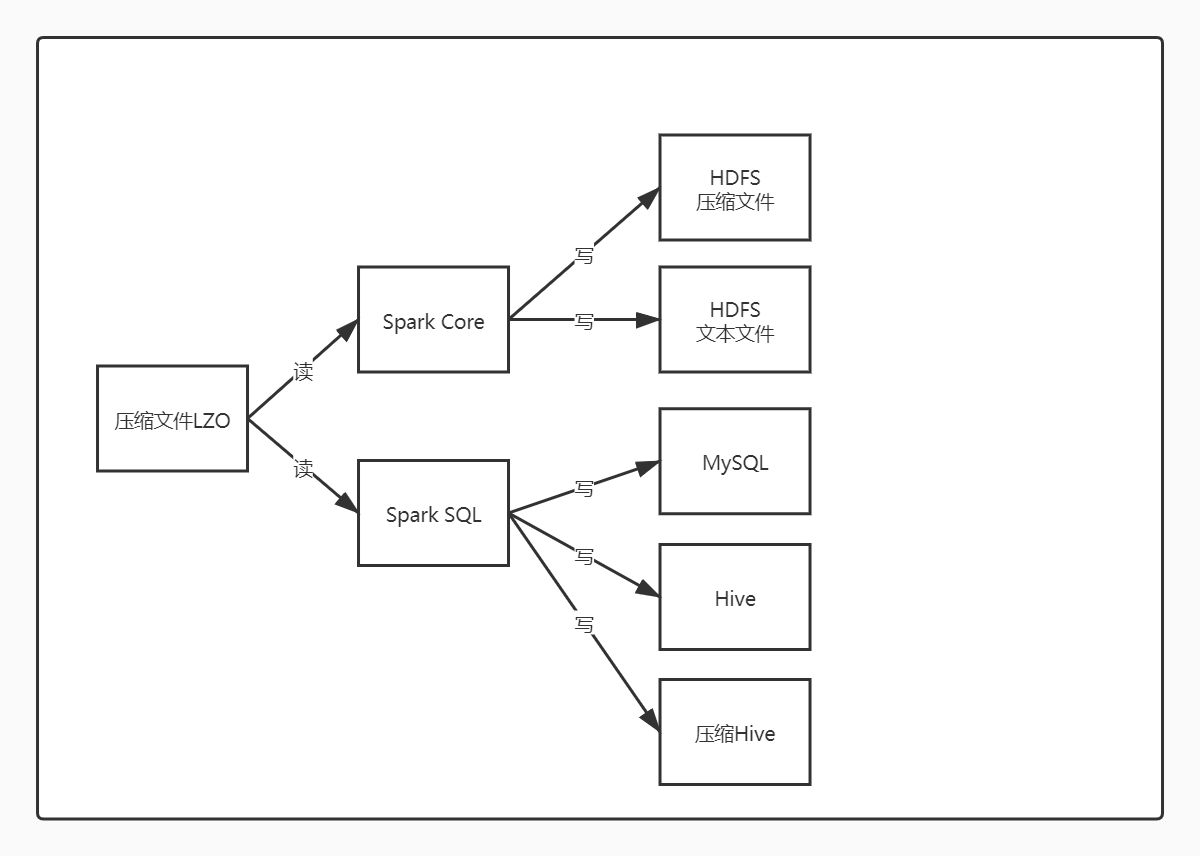
下图是我们想要做的事情
各种压缩格式的性能比较
| 压缩格式 | 工具 | 算法 | 扩展名 | 是否支持分割 | Hadoop编码/解码器 | hadoop自带 |
|---|---|---|---|---|---|---|
| DEFLATE | N/A | DEFLATE | .deflate | No | org.apache.hadoop.io.compress.DefalutCodec | 是 |
| gzip | gzip | DEFLATE | .gz | No | org.apache.hadoop.io.compress.GzipCodec | 是 |
| bzip2 | bzip2 | bzip2 | .bz2 | yes | org.apache.hadoop.io.compress.Bzip2Codec | 是 |
| LZO | Lzop | LZO | .lzo | yes(建索引) | com.hadoop.compression.lzo.LzoCodec | 是 |
| LZ4 | N/A | LZ4 | .lz4 | No | org.apache.hadoop.io.compress.Lz4Codec | 否 |
| Snappy | N/A | Snappy | .snappy | No | org.apache.hadoop.io.compress.SnappyCodec | 否 |
压缩比:Snappy<LZ4<LZO<GZIP<BZIP2
各种压缩格式的性能优缺点
| 压缩格式 | 特点 |
|---|---|
| lzo | 优点:压缩比在四种压缩方式中较高;hadoop本身支持,在应用中处理gzip格式的文件就和直接处理文本一样;有hadoop native库;大部分linux系统都自带gzip命令,使用方便 缺点:不支持split |
| snappy | 优点:压缩/解压速度也比较快,合理的压缩率;支持split,是hadoop中最流行的压缩格式;支持hadoop native库;需要在linux系统下自行安装lzop命令,使用方便 缺点:压缩率比gzip要低;hadoop本身不支持,需要安装;lzo虽然支持split,但需要对lzo文件建索引,否则hadoop也是会把lzo文件看成一个普通文件(为了支持split需要建索引,需要指定inputformat为lzo格式) |
| gzip | 优点:压缩速度快;支持hadoop native库 缺点:不支持split;压缩比低;hadoop本身不支持,需要安装;linux系统下没有对应的命令 |
| bzip2 | 优点:支持split;具有很高的压缩率,比gzip压缩率都高;hadoop本身支持,但不支持native;在linux系统下自带bzip2命令,使用方便 缺点:压缩/解压速度慢;不支持native |
读LZO文件写HDFS
需要注意的是在Spark中读取HDFS上的压缩文件,需要使用newAPIHadoopFile接口,并且传入LzoTextInputFormat,这个依赖不好解决,仓库是twitter的,下载不了,最好的办法是去github下载源码,install到本地
<dependency> |
读取文件的具体代码
val linesRDD: RDD[String] = sc.newAPIHadoopFile(in, classOf[LzoTextInputFormat], classOf[LongWritable], classOf[Text]) .map(x => x._2.toString) |
现在我们已经读进来了,可以实现我们的业务逻辑,那么最后在Spark Core中如何写出去呢? 只需要使用saveAsTextFile
etl(linesRDD,hashMap) .filter(x => x.responseSize != 0).saveAsTextFile(out) |
读LZO文件写HDFS也为LZO文件
读LZO文件上面的方法同样适用,而写为LZO文件只需要在saveAsTextFile中指定LzopCodec即可,适用于所有的格式压缩
etl(linesRDD,hashMap) .filter(x => x.responseSize != 0).saveAsTextFile(out,classOf[LzopCodec]) |
读LZO文件写MYSQL
读写表和读写文件不同,读写表更适合用Spark SQL来实现,现在我们的表的字段非常多,不适用于使用tuple的实现方式,从而自定义外部数据源,在TableScan中也是实现与上面相同代码,就能将lzo文件分片读写
override def buildScan(): RDD[Row] = { |
我在这里尝试了压缩写和普通写,都可以查询到数据
val properties = new Properties() |
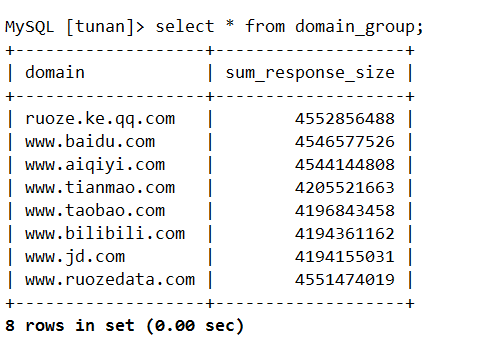
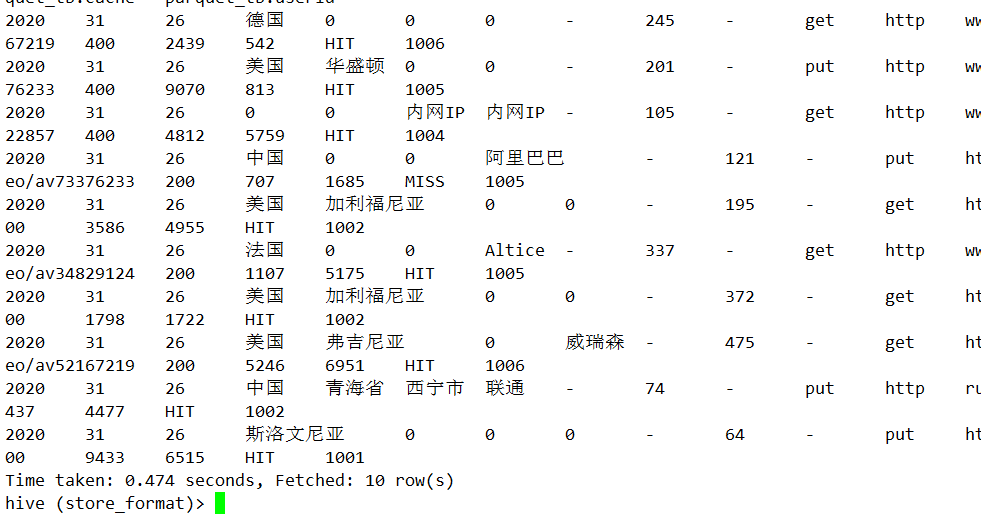
查询:
读LZO文件写HIVE
读的方式也是一样的,使用外部数据源的方式直接得到DataFrame,在写的时候指定了存储格式,而不指定压缩格式的话,会默认指定压缩格式为Snappy
textDf.write.format("parquet").mode("overwrite").saveAsTable("store_format.parquet_tb") |
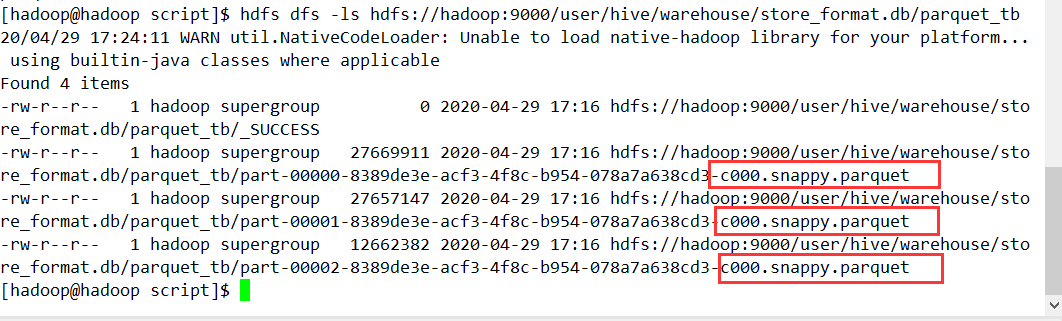
读LZO文件压缩写HIVE
读Lzo的方式如上,在这里我们尝试了几种格式的压缩写
指定了存储格式为parquet,压缩格式为lzo
textDf.write.format("parquet").option("compression","lzo").mode("overwrite").saveAsTable("store_format.parquet_tb")
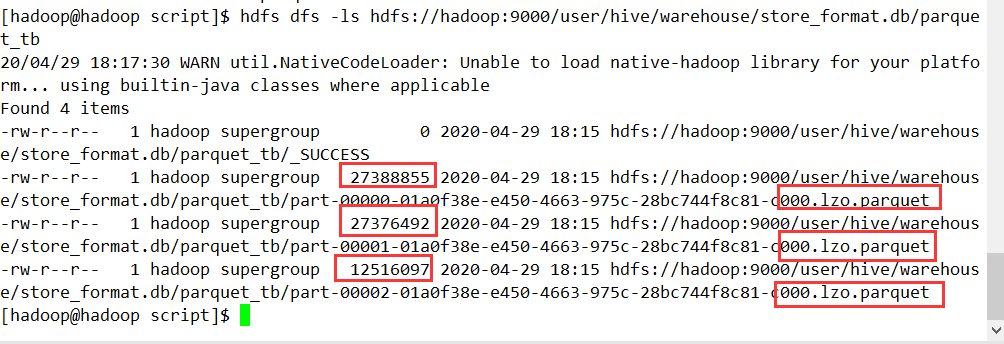
可以看得到压缩格式和存储格式发生了变化,我们继续查表
现在我们将压缩格式改为orc,存储格式还是使用lzo
textDf.write.format("orc").option("compression","lzo").mode("overwrite").saveAsTable("store_format.parquet_tb")
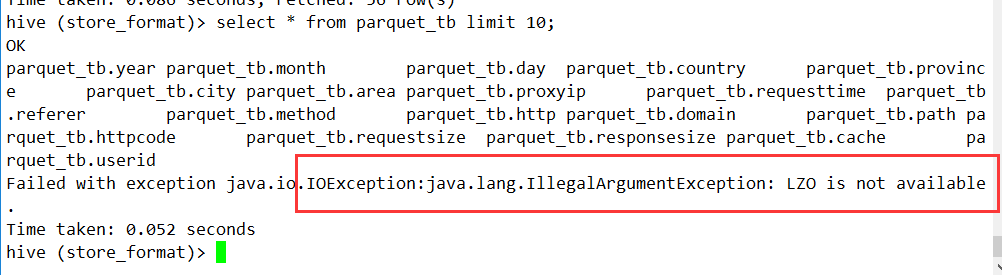
我们发现orc和lzo格式并不能一起使用,查出来的是无效的结果
继续使用压缩格式为orc,存储格式改为snappy
textDf.write.format("orc").option("compression","snappy").mode("overwrite").saveAsTable("store_format.parquet_tb")
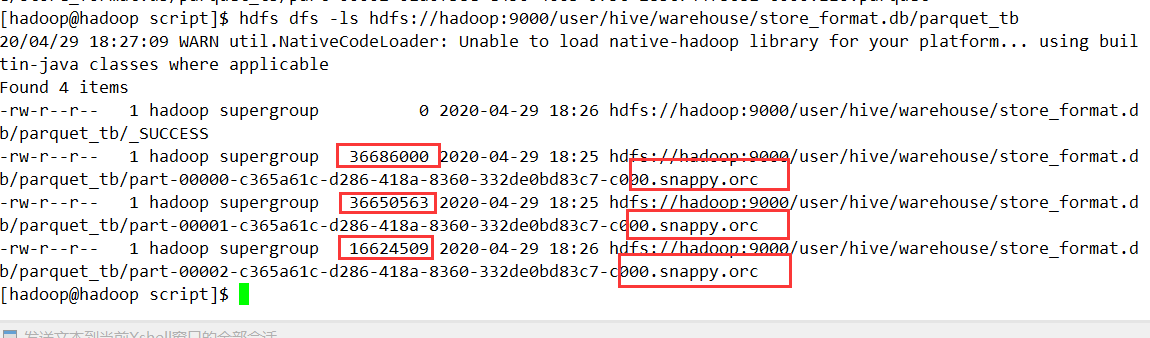
查询结果:

总结
- 使用Lzo压缩的压缩比相比于Snappy较高
- 无论哪种压缩格式在MySQL中都是无效的
- Parquet可以和Snappy或者Lzo搭配使用,ORC可以和Snappy或者Bzip搭配使用
- ORC不能和Lzo搭配使用
